COVID-19 und Todesziffern
In den Jahren 2020 und 2021 wurde die gesamte Weltbevölkerung regelmässig über die laufende Anzahl Todesfälle und laufende Anzahl Ansteckungen von COVID-19 informiert. Dies in einen breiteren Kontext zu setzen wurde für die Bevölkerung etwas erschwert, da nur selten Todesfälle der vergangenen Jahren gezeigt und noch nie zuvor den Verlauf von viraler RNA/DNA (+ssRNA im Fall von COVID-19) in der Bevölkerung so genau ermittelt wurde.
Schauen wir uns doch in dem Fall mal die Todesfälle seit 1803 an - zum Glück wurden diese Daten vom Schweizerischen Bundesamt für Statistik regelmässig erhoben und können für jedermann einfach runtergeladen werden.
Hierfür benutzen wir hier die open source software R und R studio. Natürlich könnte man die Daten auch mit anderen Programmen anschauen, aber R verfügt über interessante features wie interaktive Darstellungen (z.B. shiny apps) und verschiedene Pakete wie z.B. das vom Bundesamt für Statistik (BFS) für einfaches Runterladen und Bearbeiten von Datenbanken.
Die untensteheden R code Abschnitte erlauben über einfaches copy-paste das Runterladen der Datensätze, Rausfiltern und Darstellen der Daten die uns interessieren. Also schnell, kompakt und reproduzierbar (Stand 10.6.2021 - an stellen wo nur 1 Datenpunkt pro Jahr/Monat vorhanden ist, wählen wir ein arbiträrer Tag für die Darstellungen)
library(tidyverse)
BFS::bfs_get_dataset(url_px = "https://www.bfs.admin.ch/bfsstatic/dam/assets/16664926/master") %>%
filter(demografisches_merkmal_und_indikator=='Todesfälle - Total') %>%
mutate(jahr=lubridate::dmy(glue::glue('1506{jahr}'))) %>%
filter(!is.na(value), !is.na(jahr)) %>%
ggplot(aes(x=jahr, y=value)) + geom_line() + theme_classic() + labs(y='Jährliche Anzahl Tote')
bfs_get_dataset(url_px="https://www.bfs.admin.ch/bfsstatic/dam/assets/16664926/master") %>% mutate(jahr=lubridate::dmy(glue::glue('1506{jahr}'))) %>%
dplyr::filter(demografisches_merkmal_und_indikator=='Todesfälle je 1000 Einwohner', !is.na(value)) %>%
ggplot(aes(x=jahr, y=value)) + geom_line() + theme_classic() + labs(x="Jahr", y="Jährliche Anzahl Tote pro 1000 Einwohner")BFS::bfs_get_dataset(url_px="https://www.bfs.admin.ch/bfsstatic/dam/assets/16664926/master") %>% mutate(Datum=case_when(
demografisches_merkmal_und_indikator=='Todesfälle im Januar' ~ lubridate::dmy(glue::glue('1501{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im Februar' ~ lubridate::dmy(glue::glue('1502{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im März' ~ lubridate::dmy(glue::glue('1503{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im April' ~ lubridate::dmy(glue::glue('1504{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im Mai' ~ lubridate::dmy(glue::glue('1505{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im Juni' ~ lubridate::dmy(glue::glue('1506{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im Juli' ~ lubridate::dmy(glue::glue('1507{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im August' ~ lubridate::dmy(glue::glue('1508{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im September' ~ lubridate::dmy(glue::glue('1509{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im Oktober' ~ lubridate::dmy(glue::glue('1510{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im November' ~ lubridate::dmy(glue::glue('1511{jahr}')),
demografisches_merkmal_und_indikator=='Todesfälle im Dezember' ~ lubridate::dmy(glue::glue('1512{jahr}'))
)) %>% dplyr::filter(!is.na(value), !is.na(Datum)) %>%
ggplot(e3,aes(x=jahr, y=Anzahl_Todesfalle)) +
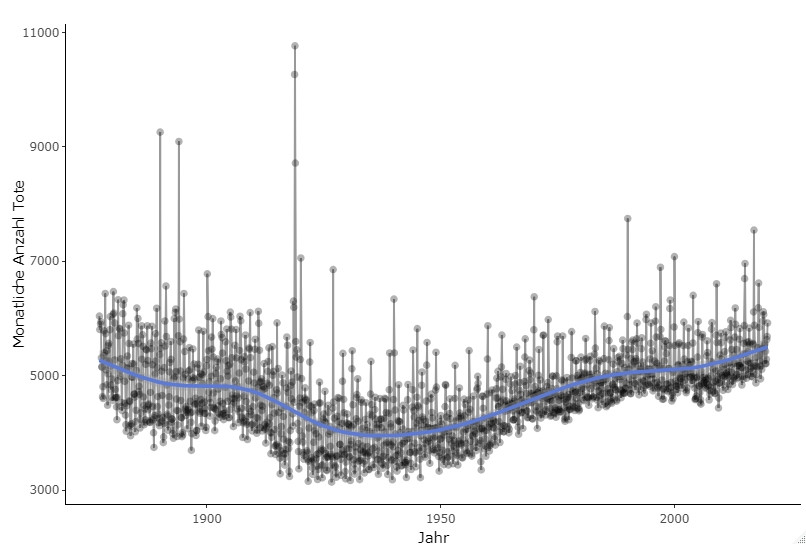
geom_point(alpha=0.3) + geom_line(alpha=0.4) + geom_smooth() + theme_classic() + labs(x="Jahr", y='Monatliche Anzahl Todesfälle')
Obige Daten zeigen uns leider nur vergangene Jahr bis und mit 2019. Schauen wir uns nun aber noch die aktuellen Zahlen an. Diese können hier runtergeladen werden.
dat = vroom::vroom('<Speicherort 1>') %>% dplyr::select(endend, Alter, AnzTF_HR) %>% dplyr::filter(!is.na(AnzTF_HR)) %>%
dplyr::rename("Endend"="endend", "Anzahl_Todesfalle"="AnzTF_HR")vroom::vroom('<Speicherort 2>') %>% mutate(Anzahl_Todesfalle=as.numeric(Anzahl_Todesfalle)) %>%
dplyr::filter(!is.na(Anzahl_Todesfalle)) %>% dplyr::select(Endend, Alter, Anzahl_Todesfalle) %>% rbind.data.frame(dat) %>%
dplyr::filter(Anzahl_Todesfalle!='.') %>% mutate(date=as.POSIXct(lubridate::fast_strptime(Endend, format = '%d.%m.%Y')), Alter=as.factor(Alter), Anzahl_Todesfalle=as.numeric(Anzahl_Todesfalle),
demografisches_merkmal_und_indikator= 'Todesfälle 2010-2021') %>%
ggplot(e4, aes(x=jahr, y=Anzahl_Todesfalle, color=Alter)) + geom_line() + theme_classic() + labs(x="Jahr", y='Todesfälle 2010-2021')
Kommentare oder Fragen sind über discuss.analysed.ch (open source software rocketchat) im Channel #covid19 jederzeit sehr willkommen.

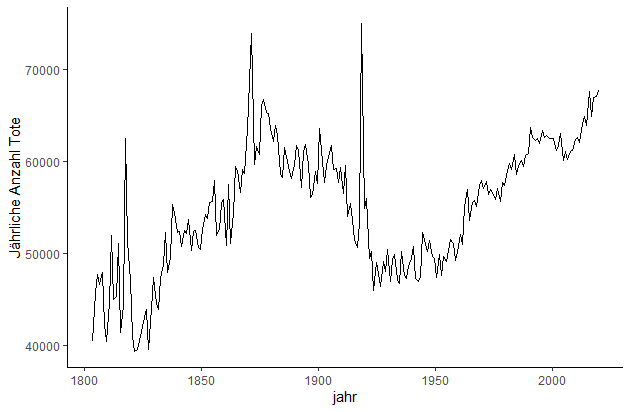
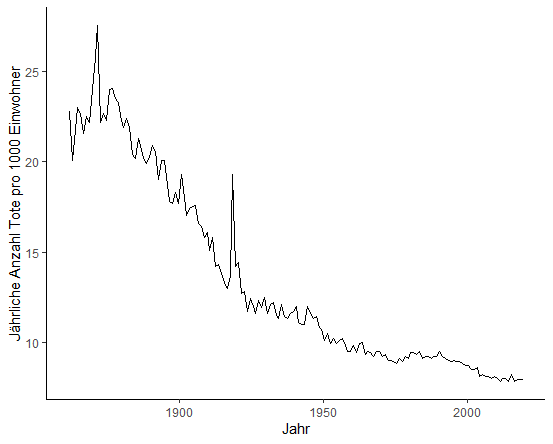
No Comments